Havainnon spesifisyys
Useimmissa tapauksissa alukkeen suunnittelun tarkoituksena on maksimoida PCR:n spesifisyys.Tämä määräytyy monien muuttujien enemmän tai vähemmän ennakoitavan vaikutuksen perusteella.Yksi tärkeä muuttuja on sekvenssi alukkeen 3'-päässä.
Tärkeää on, että spesifisyyteen suunnitellut PCR-määritykset säilyttävät todennäköisemmin korkean tehokkuuden laajalla dynaamisella alueella, koska määritys ei tuota epäspesifisiä monistustuotteita, jolloin ne kilpailevat PCR-reagenssien kanssa tai inhiboivat pääamplifikaatioreaktion.
Tietysti joissain tapauksissa spesifisyys ei ole tärkeintä, esimerkiksi kun tavoitteena on kvantifioida läheisesti toisiinsa liittyviä, mutta erilaisia taudinaiheuttajia, tarvitaan erityisiä suunnittelu-, optimointi- ja todentamisstandardeja.
Sulamiskäyrä on standardimenetelmä amplikonien spesifisyyden arvioimiseksi, ainakin sen suhteen, onko amplifioitu yksittäinen kohde.On kuitenkin korostettava, että sulamiskäyrät voivat olla harhaanjohtavia, koska niihin voivat vaikuttaa esimerkiksi suboptimaalisten alukkeiden ja alhaisten templaattipitoisuuksien yhteisvaikutukset.
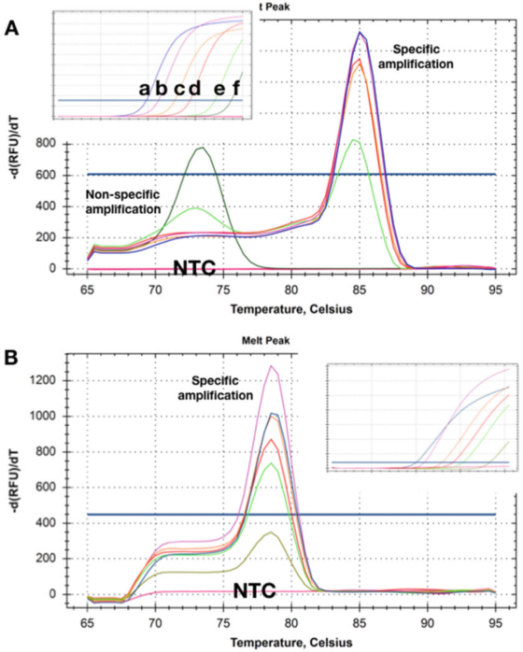
P5 |Sulamiskäyrä näyttää Tm-siirtymät, jotka on saatu kahdesta kahden kohde-DNA:n eri määrien havaitsemisesta.
A. Suuremmilla pitoisuuksilla (ad)) ei ole selvää alukedimeeriä sen jälkeen, kun qPCR-mittaus on suoritettu.Kun templaattipitoisuus laskee 50 kopioon (e), epäspesifinen tuote alkaa ilmestyä ja siitä tulee ainoa tuote pienimmällä pitoisuudella (f).
B. Testi rekisteröi samat Tms:t kaikilla tavoitepitoisuuksilla, eikä ilmeistä alukedimeeriä ollut edes pienimmällä pitoisuudella (5 kopiota).Näitä kahta tunnistusmenetelmää käytettäessä NTC:issä ei havaittu amplifikaatiotuotteita.
P5 esittää liukenemiskäyrät, jotka on saatu näytteistä, joissa templaattia on läsnä eri pitoisuuksina.P 5a osoittaa, että kahdella pienimmällä pitoisuudella tuotettujen epäspesifisten monistustuotteiden Tms ovat alhaisemmat kuin spesifisten amplikonien.
Ilmeisesti tätä havainnointimenetelmää ei voida käyttää luotettavasti havaitsemaan kohteita, joita esiintyy pieninä pitoisuuksina.
Mielenkiintoista on, että NTC:t, eli näytteet, joissa ei ole lainkaan DNA:ta, eivät tallentaneet (epäspesifisiä) monistustuotteita, mikä osoittaa, että genominen tausta-DNA voi osallistua epäspesifiseen monistumiseen/polymerointiin.
Joskus tällaisia taustaalukkeita ja epäspesifistä monistumista ei voida korjata, mutta usein on mahdollista suunnitella havaitsemismenetelmä, jolla ei ole epäspesifistä monistumista missään templaattipitoisuudessa ja NTC:ssä (P 5b).
Tässä jopa tavoitepitoisuuden vahvistumisen kirjaaminen Cq:lla 35 tuottaa erityisen liukenemiskäyrän.Samoin NTC:t eivät osoittaneet merkkejä epäspesifisestä vahvistumisesta.Joskus havaitsemiskäyttäytyminen voi olla riippuvainen emäliuoksesta, ja tietyissä puskurikoostumuksissa havaitaan vain epäspesifistä monistumista, mikä voi liittyä erilaisiin Mg2+-konsentraatioihin.
Havainnon vakaus
Ta:n optimointi on hyödyllinen vaihe qPCR-detektion empiirisessä verifiointi- ja optimointiprosessissa.Se antaa suoran osoituksen alukesarjan kestävyydestä näyttämällä lämpötilan (tai lämpötila-alueen), joka tuottaa alhaisimman Cq:n vahvistamatta NTC:tä.
Kaksi-neljänkertainen herkkyysero ei ehkä ole tärkeä ihmisille, joilla on korkea mRNA:n ilmentyminen, mutta diagnostisissa testeissä se voi tarkoittaa eroa positiivisten ja väärien negatiivisten tulosten välillä.
QPCR-alukkeiden Ta-ominaisuudet voivat vaihdella suuresti.Jotkut testit eivät ole kovin kestäviä, ja jos niitä ei suoriteta alukkeiden optimaalisen Ta-arvon alapuolella, ne romahtavat nopeasti.
Tämä on tärkeää, koska tämäntyyppinen havaitseminen on usein ongelmallista todellisessa maailmassa, ja näytteen puhtaus, DNA:n pitoisuus tai muun DNA:n läsnäolo ei ehkä ole optimaalinen.
Lisäksi tavoitekopiomäärä voi vaihdella laajalla alueella, ja reagenssit, muovivälineet tai instrumentit voivat olla erilaisia kuin testin asettamisessa käytetyt.
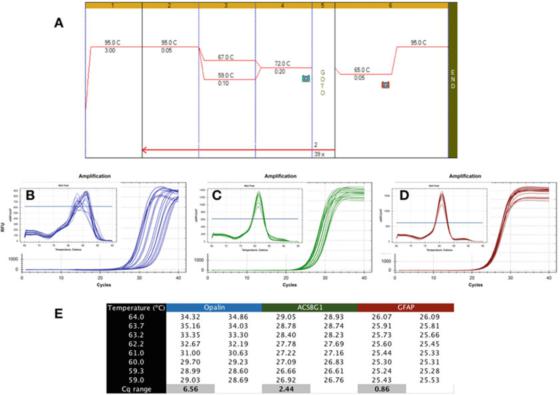
P6|Lämpötilagradientti osoittaa PCR-tunnistuksen erilaisen vahvuuden.
A. Käytä Biolinen Sensifast SYBR mastermixiä (luettelonumero BIO-98050) PCR:n suorittamiseen ihmisen aivojen RNA:sta valmistetulle cDNA:lle.
B. Käytä Bio-Radin CFX qPCR -laitetta tallentaaksesi apaleenin monistuskartan ja liukenemiskäyrän (NM_033207, F: GCCATGGAGGAAAGTGACAGACC, R: CTCATGTGTGGGTGATCTCCTAGG).
C. ACSBG1:n monistuskäyrä ja sulamiskäyrä (NM_015162.4, F: CTACACTTCCGGCACCACTGG, R: GTCCACGTGATATTGTCTTGACTCAG).
D. GFAP:n monistuskäyrä ja liukenemiskäyrä (NM_002055.5, F: TGGAGAGGAAGATTGAGTCGCTGG, R: CGAACCTCCTCCTCGTGGATCTTC).
E. Cq:t, jotka on tallennettu eri hehkutuslämpötiloissa, osoittaen eron Cq:ssa, joka on tallennettu 7 °C:n lämpötilagradientin alla.
P 6 näyttää tyypillisen tuloksen ei-toivotusta testistä, jossa qPCR suoritettiin käyttämällä Tas-gradienttia välillä 59C ja 67C (P 6a), käyttäen alukkeita kolmelle ihmisen aivospesifiselle geenille.
Amplifikaatiokaaviosta voidaan nähdä, että Opalin-alukkeet ovat kaukana ihanteellisista, koska niiden optimaalinen Ta-alue on hyvin kapea (kuva 6b), eli Cq:t ovat laajalti hajallaan, mikä johtaa siihen, että Cqs on merkittävästi verrattuna niiden optimaaliseen Cqs Low -arvoon.
Tämä tunnistusmenetelmä on epävakaa ja voi johtaa suboptimaaliseen vahvistukseen.Siksi tämä alukepari tulisi suunnitella uudelleen.Lisäksi sulamiskäyräanalyysi (inset) osoittaa, että tämän tunnistusmenetelmän spesifisyys voi myös olla ongelmallinen, koska kunkin Ta:n sulamiskäyrä on erilainen.
P 6c:ssä esitetty ACSBG1-tunnistusmenetelmä on vankempi kuin yllä oleva Opalin-tunnistusmenetelmä, mutta se on silti kaukana ihanteellisesta, ja on todennäköistä, että sitä voidaan parantaa.
Korostamme kuitenkin, että robustisuuden ja spesifisyyden välillä ei ole välttämätöntä yhteyttä, koska tällä tunnistusmenetelmällä tuotettu liukenemiskäyrä näyttää saman huippuarvon kaikissa Tasoissa (inset).
Toisaalta kestävyystesti on paljon siedettävämpi ja tuottaa samanlaisia Cq:itä laajalla Tas-alueella, kuten P 6d:ssä esitetyssä GFAP-testissä.
Samalla 8 Celsius-asteen alueella saatu Cqs-ero on pienempi kuin 1, ja liukenemiskäyrä (inset) vahvistaa havaitsemisominaisuudet tällä lämpötila-alueella.On syytä huomata, että laskettu Tas ja todellinen Ta-alue voivat olla hyvin erilaisia.
On olemassa monia ohjeita, jotka on suunniteltu auttamaan tutkijoita suunnittelemaan tehokkaita alukkeita, joista suurin osa perustuu pitkään vakiintuneisiin sääntöihin ja paljon huomiota on kiinnitetty alukkeiden 3′päähän.On usein suositeltavaa sisällyttää G tai C 3'-päähän ja kaksi G- tai C-kantaa (GC-kiinnike), mutta enintään kaksi viimeisestä viidestä emäksestä.
Käytännössä nämä säännöt voivat ohjata tutkijoita, mutta ne eivät välttämättä ole oikeita kaikissa olosuhteissa.
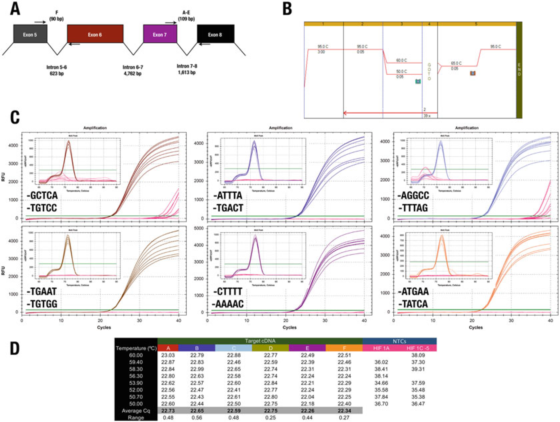
P7 |Alukkeen 3′-päällä on vain vähän vaikutusta spesifisyyteen tai tehokkuuteen.
A. Ihmisen HIF-1a (NM_181054.2) -geenin alukkeiden sijainti.
B. Käytä Agilent Brilliant III SYBR Green -emäliuosta (luettelonro 600882) kuuden testikohteen vahvistamiseen.
C. Bio-Radin CFX qPCR -laitteella ja 3'-pään alukkeilla tallennettu monistuskäyrä ja sulamiskäyrä.NTC:t näkyvät punaisella.
D. Cqs-tietue jokaisesta testikappaleesta
Esimerkiksi P 7:n tulos on ristiriidassa 3′-säännön kanssa.Kaikki mallit tuottavat periaatteessa samat tulokset, ja vain kaksi alukeyhdistelmää johtaa epäspesifiseen monistukseen NTC:ssä.
Emme kuitenkaan voi tukea GC-leikkeen vaikutusta, koska tässä tapauksessa A:n tai T:n käyttäminen enintään 30 emäksenä ei vähennä spesifisyyttä.
Testi C, jossa F-aluke päättyy GGCC:hen, tallensi Cq:t NTC:issä, mikä osoittaa, että näitä sekvenssejä saatetaan haluta välttää 30-päässä.Korostamme, että ainoa tapa määrittää alukeparin paras 3'-pään sekvenssi on arvioida joitain ehdokasalukkeita kokeellisesti.
Vahvistuksen tehokkuus
Tärkeää on, että vaikka epäspesifinen PCR-detektio ei koskaan voi tulla spesifiseksi, amplifikaatiotehokkuutta voidaan säätää ja maksimoida monin eri tavoin muuttamalla entsyymiä, emäliuosta, lisäaineita ja sykliolosuhteita.
PCR-detektion tehokkuuden arvioimiseksi on parasta käyttää sarjalaimennusta, joka on 10 tai 5 kertaa kohdenukleiinihappo, eli "standardikäyrämenetelmää".
Jos PCR-amplikoneja tai synteettisiä DNA-kohteita käytetään luomaan standardikäyrä, näiden kohteiden sarjalaimennokset tulee sekoittaa vakiomäärään tausta-DNA:ta (kuten genominen DNA:ta).
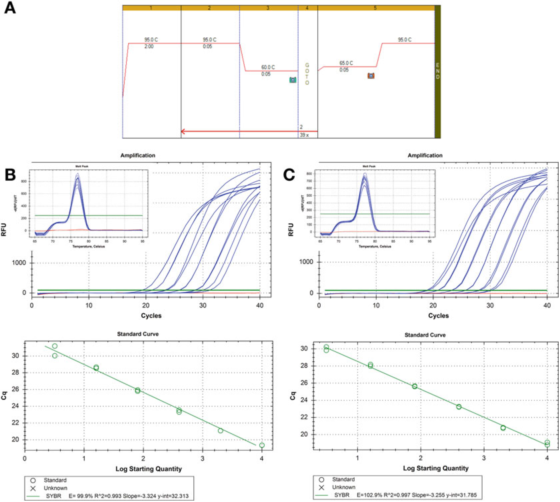
P8 |Laimennuskäyrä PCR:n tehokkuuden arvioimiseksi.
A. Käytä HIF-1:n alukkeita: F: AAGAACTTTTAGGCCGCTCA ja R: TGTCCTGTGGTGACTTGTCC ja Agilent's Brilliant III SYBR Green mastermixiä (luettelonumero 600882) PCR- ja sulamiskäyräolosuhteisiin.
B. 100 ng RNA:ta käänteistranskriptoitiin, laimennettiin 2 kertaa ja sarjalaimennetut cDNA-näytteet laimennettiin 5 kertaa 1 ng:ksi ihmisen genomista DNA:ta.Sulamiskäyrä näkyy sisäpuolella.
C. RT-reaktio, laimennus ja sarjalaimennus toistettiin toiselle cDNA-näytteelle, ja tulokset olivat samanlaiset.
P 8 esittää kaksi standardikäyrää, käyttäen samaa havainnointimenetelmää kahdella eri cDNA-näytteellä, tulos on sama tehokkuus, noin 100%, ja myös R2-arvo on samanlainen, eli sovitusaste koetietojen ja regressioviivan tai datan lineaarisuusaste välillä.
Kaksi vakiokäyrää ovat vertailukelpoisia, mutta eivät täsmälleen samat.Jos tarkoitus on kvantifioida kohde tarkasti, on huomioitava, että kopiomäärälaskelman toimittaminen ilman epävarmuuden selittämistä ei ole hyväksyttävää.
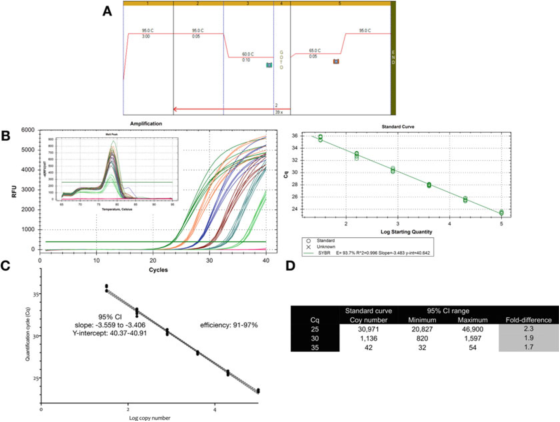
P9 |Mittauksen epävarmuus, joka liittyy kvantifiointiin standardikäyrällä.
A. Käytä alukkeita GAPDH:lle (NM_002046) PCR:n ja sulamiskäyrän olosuhteiden suorittamiseen.F: ACAGTTGCCATGTAGACC ja R: TAACTGGTTGAGCACAGG ja Biolinen Sensifast SYBR mastermix (luettelonumero BIO-98050).
B. Monistumiskaavio, sulamiskäyrä ja standardikäyrä tallennettu Bio-Radin CFX qPCR -laitteella.
C. Vakiokäyräkaavio ja 95 %:n luottamusväli (CI).
D. Laimennuskäyrästä johdettujen kolmen Cq-arvon kopiomäärä ja 95 %:n luottamusväli.
P 9 osoittaa, että optimoidun testin tapauksessa yksittäisen standardikäyrän luontainen vaihtelevuus on noin 2 kertaa (95 %:n luottamusväli, minimistä maksimiin), mikä voi olla pienin odotettavissa oleva vaihtelu.
Aiheeseen liittyvä tuote:
Animal Tissue Direct PCR -pakkaus
Postitusaika: 30.9.2021